近年来,空间转录组技术(Spatial Transcriptomics, ST)迅速发展,为探索细胞功能、调控机制及微环境互作提供了前所未有的空间视角。它被 Nature Methods 评为2020年度技术(Method of the Year 2020),并被誉为“将彻底改变细胞生物学、病理学及组织学的研究方式”。
空间转录组分类
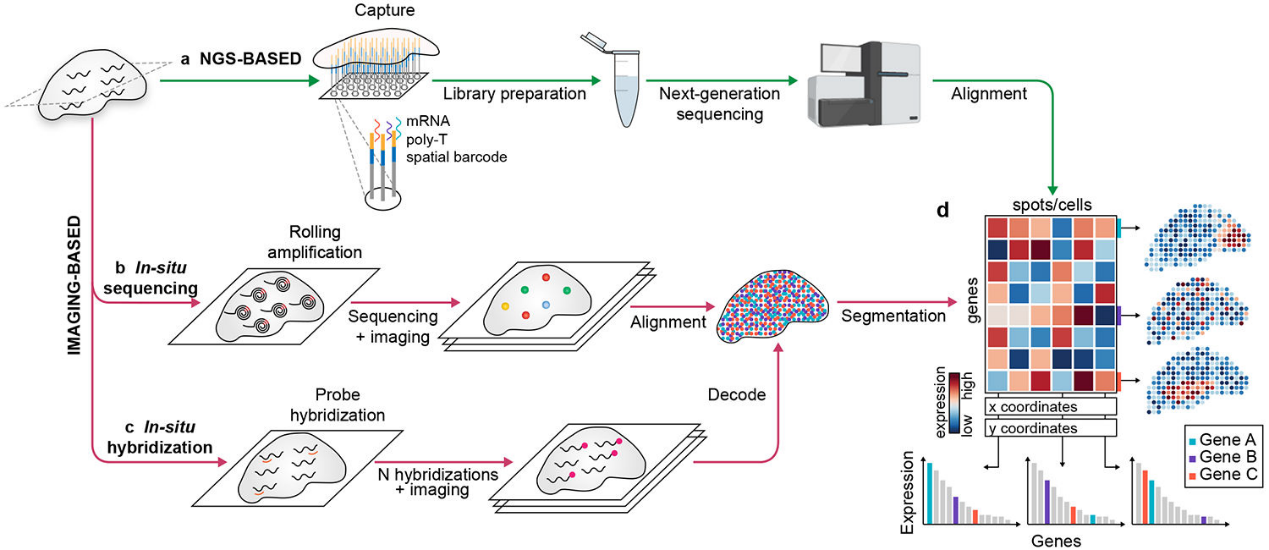
空间转录组结合高通量转录组测序与组织结构空间信息,弥补了传统单细胞RNA测序(scRNA-seq)在空间定位上的缺陷。ST技术主要分为两类:
- 测序型 ST(sST):通过空间条形码阵列捕获RNA,实现空间分辨测序(图1a);
- 成像型 ST(iST):基于荧光探针与循环成像,在单分子分辨率下实现靶基因的原位测序(图1b)和原位杂交(图1c)。
图1. 空间转录组主要分类1
为什么要关注 DBiT-seq? 它是 ST 阵营中 唯一通过微流控双条码就能实现 RNA-蛋白-表观组学 同片读取、且像素可低至 10 µm 的技术——既保留了近单细胞分辨率,又无需昂贵的高端成像系统。
DBiT-seq 方法概览2
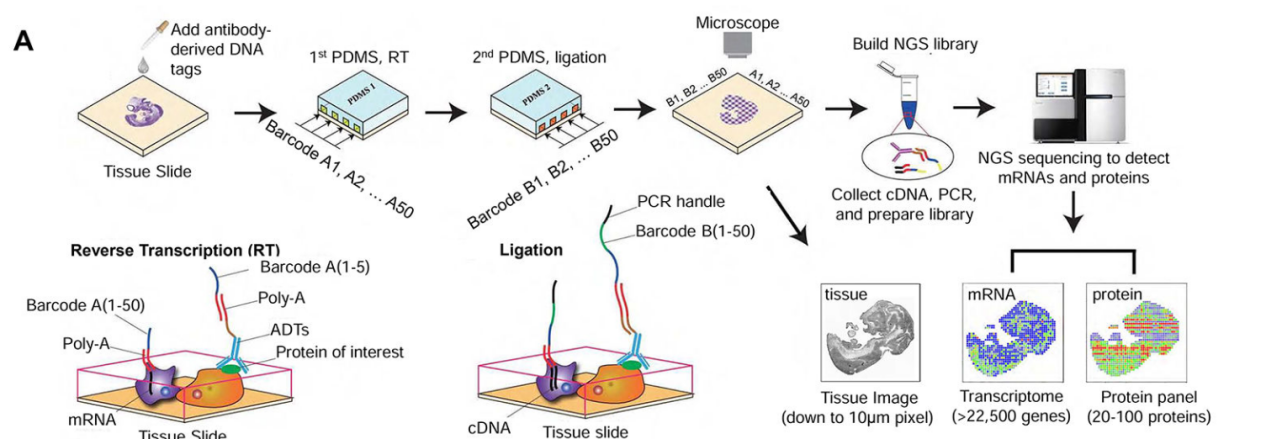
样本前处理:对新鲜或 FFPE 组织切片进行化学固定并轻度渗透,使 RNA / 蛋白原位稳定,同时打开细胞膜通道,确保后续条码溶液能够均匀进入组织。
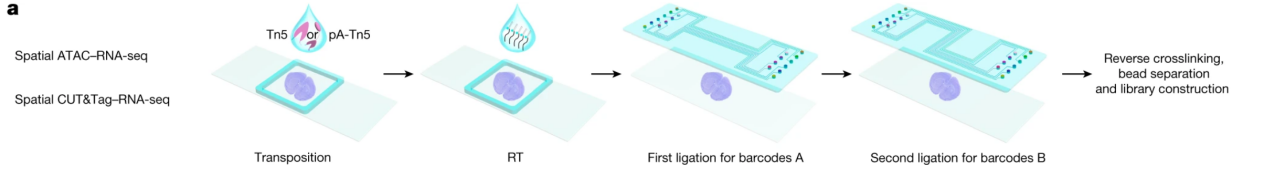
第一次注入(X-Chip):PDMS 芯片含 50 条平行微通道(10–50 µm 宽)。负压 将 条码 A(oligo-dT + UMI + 50 nt 接头)抽入;条码 A 在切片表面与 poly(A)+ mRNA 退火并就地逆转录 → 形成 条码A-cDNA 条带。
第二次注入(Y-Chip):第二块芯片垂直放置;条码 B(带磷酸化 5' 端)被同样的负压抽流;在两条道交叉点处,T4 DNA ligase 将条码 B 与条码A-cDNA 3' 接头连接;这样每一个交点 (= 像素) 获得唯一 “A_i-B_j” 组合。;
二维像素网格:A×B 组合数即像素数 (10x10、20x20 或 50×50);矩阵坐标由通道序号天然标定。
逆交联 & 裂解回收:采用专用缓冲液解除固定交联并裂解组织,将携带坐标信息的 cDNA、抗体-DNA 或表观学片段释放到溶液中,为建库做准备。
文库制备与上机:根据目标组学(RNA、蛋白-ADT、ATAC、CUT&Tag 等)选择相应建库方案,扩增并纯化片段后进行高通量测序。
图2. DBiT实验流程
关键参数
| 指标 | 参数 | 应用价值 |
| 通道宽度 | 10 µm / 20 µm / 50 µm | 10 µm 逼近单细胞,50 µm 适配大组织 |
| 检测面积 | 1×1 mm²、2×2 mm²、5×5 mm² | 胚胎 → 器官 → 切片 全覆盖 |
| 可测组学 | RNA、蛋白、ATAC、CUT&Tag | 一套硬件,多模态扩展 |
所有条码寡核苷酸开放自定义。
标准实验流程
| 步骤 | 核心 | 目的与要点 |
| 切片固定/渗透 | 锁定组织形态、打开通道 | 保证 RNA / 蛋白原位保真,具备条码进入条件 |
| X-Chip 条码 A | 横向编码+原位逆转录 | 真空抽液,完成第一维编码,cDNA 获得 A 坐标 |
| Y-Chip 条码 B | 纵向编码+连接反应 | 垂直注入第二维条码,像素获得唯一 A×B 组合,二维坐标完成 |
| 逆交联 / 裂解 | 释放条码-分子复合物 | 带坐标的 cDNA 片段进入溶液 |
| 文库构建 & NGS | 转化为测序文库 | 得到可上机的 RNA / 表观等读段 |
DBiT-seq技术优势
近乎单细胞的分辨率:10 µm 通道即可逼近单细胞尺度,但仍可一次覆盖 25 mm² 大视野,兼顾精细度与全景。
真正的多组学同源:同一张切片可顺序获得 RNA、蛋白、ATAC、CUT&Tag 等信号——所有层级共用同一 XY 坐标,不再为数据对齐头疼。
条码完全开放、自主扩容:A/B 通道预留空白位,研究者可随时插入自定义寡核苷酸或抗体-DNA 标签;无需等待厂商升级 capture-slide。
流程简单、不依赖高端成像:两轮真空抽流+常规 NGS 即可完成实验;无需多轮共聚焦或超分辨显微镜,降低进入门槛。
应用案例
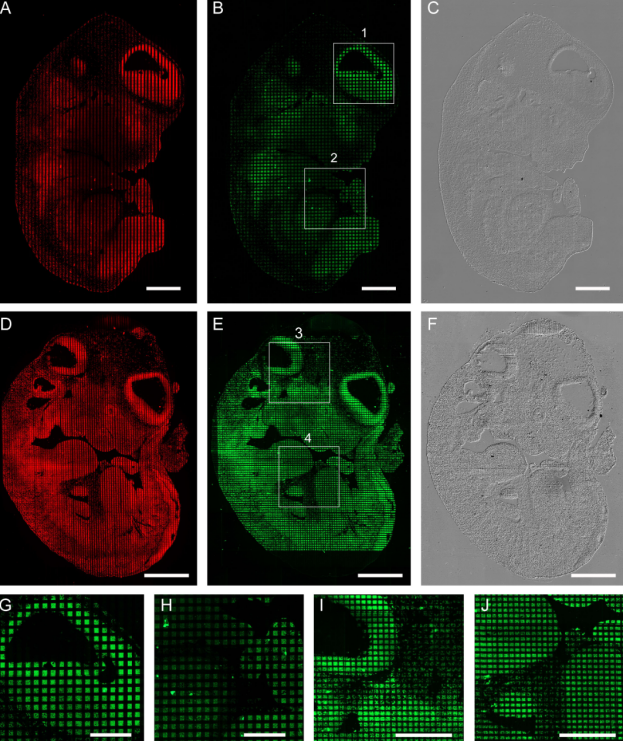
Spatial-ATAC / CUT&Tag-RNA3沿用 DBiT-seq 的二维条码编码后,先做原位逆转录捕获 RNA,再直接在同一切片进行 ATAC 或 CUT&Tag,借助同一对 A×B 坐标同时回收“开放染色质 / 组蛋白修饰”与转录组信号,实现表观-转录双模态共定位,并开发算法解析二者动态关联。结果揭示胼胝体髓鞘基因在“开放—沉默—激活”三阶段的空间演变,为后续研究阿尔茨海默病中表观驱动的斑块扩散提供了可能路径。
图3. Spatial-ATAC/CUT&Tag-RNA 联合测序揭示胼胝体髓鞘基因的空间动态3
Spatial-CITE-seq4在 DBiT-seq 的条码 A 通道中加入多重抗体-DNA(ADT)标签,使 RNA 与 189 种蛋白位点同时绑定坐标;条码 B 注入后形成像素网格,最终得到 RNA-蛋白同源图谱。分析发现 CD21⁺ 成熟 B 细胞和 CXCL13⁺ 滤泡-辅助 T 细胞存在显著空间互作,而部分趋化因子呈现“蛋白高 / mRNA 低”现象,提示转录后调控在肿瘤微环境中扮演关键角色。
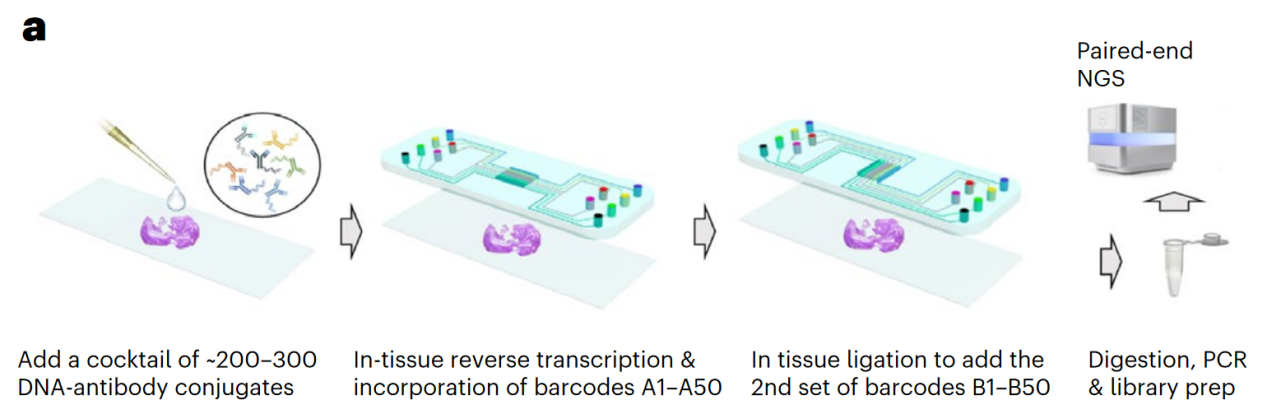 图4. Spatial-CITE-seq 在人扁桃体实现 189 种蛋白与全转录组的共定位解析4
图4. Spatial-CITE-seq 在人扁桃体实现 189 种蛋白与全转录组的共定位解析4
ARP-seq / CTRP-seq × CODEX5以 DBiT-seq 为核心先捕获 A (open chromatin)、R (RNA)、P (protein) 或 C (CUT&Tag)-T (RNA)-P (protein),再叠加 CODEX 高维成像;DBiT 的坐标条码把 ATAC/CUT&Tag-RNA-蛋白三模态与 CODEX 图像对齐,构成表观-转录-蛋白-形态四维整合平台。研究发现皮质层特异转录因子(SATB2、FOXP2)的染色质可及性呈“波浪式”向浅层传播,形成表观“时间记忆”,为神经发育时序调控提供了全新视角。
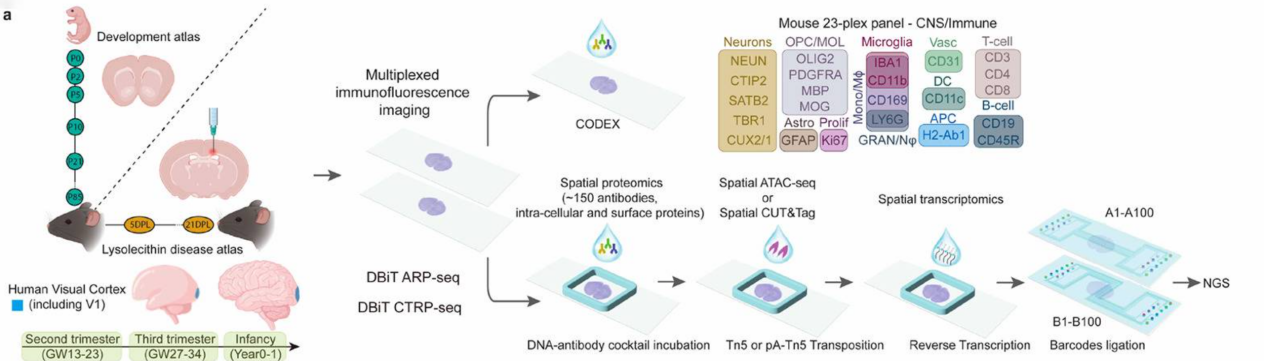 图5. ARP/CTRP-seq × CODEX 多模态解码小鼠大脑 P0–P21 发育时空图谱5
图5. ARP/CTRP-seq × CODEX 多模态解码小鼠大脑 P0–P21 发育时空图谱5
FluidicLab-空间组学的核心模块--DBiT-seq套装
在“大样本、真临床、可复制”成为新评价指标的今天,DBiT-seq 凭借 高分辨 + 多组学 + 成本友好,让空间组学从“小规模炫技”迈向“高通量常规”。FluidicLab提供的DBiT-seq系统采用压力驱动设备配合聚二甲基硅氧烷(PDMS)微流控芯片通过垂直交叉的微通道(宽度可选10 um、20 um、50 um)对组织切片进行二维编码。
图6. DBiT-seq套装
应用方向
DBiT-seq结合多重抗体-DNA 标签的 Spatial-CITE-seq,同步定位上百种蛋白并与 RNA 数据同源配准
DBiT-seq结合 Tn5/CUT&Tag 体系的 Spatial-ATAC / CUT&Tag-RNA,在同一切片绘制染色质可及性与组蛋白修饰的空间图谱
参考文献
- Rao, A., Barkley, D., França, G. S. & Yanai, I. Exploring tissue architecture using spatial transcriptomics. Nature596, 211–220 (2021).
- Liu, Y., Enninful, A., Deng, Y. & Fan, R. Spatial transcriptome sequencing of FFPE tissues at cellular level. 2020.10.13.338475 Preprint at https://doi.org/10.1101/2020.10.13.338475 (2020).
- Zhang, D. et al.Spatial epigenome–transcriptome co-profiling of mammalian tissues. Nature616, 113–122 (2023).
- Liu, Y. et al.High-plex protein and whole transcriptome co-mapping at cellular resolution with spatial CITE-seq. Nat. Biotechnol.41, 1405–1409 (2023).
- Zhang, D. et al.Spatial dynamics of mammalian brain development and neuroinflammation by multimodal tri-omics mapping. bioRxiv2024.07.28.605493 (2024) doi:10.1101/2024.07.28.605493.




























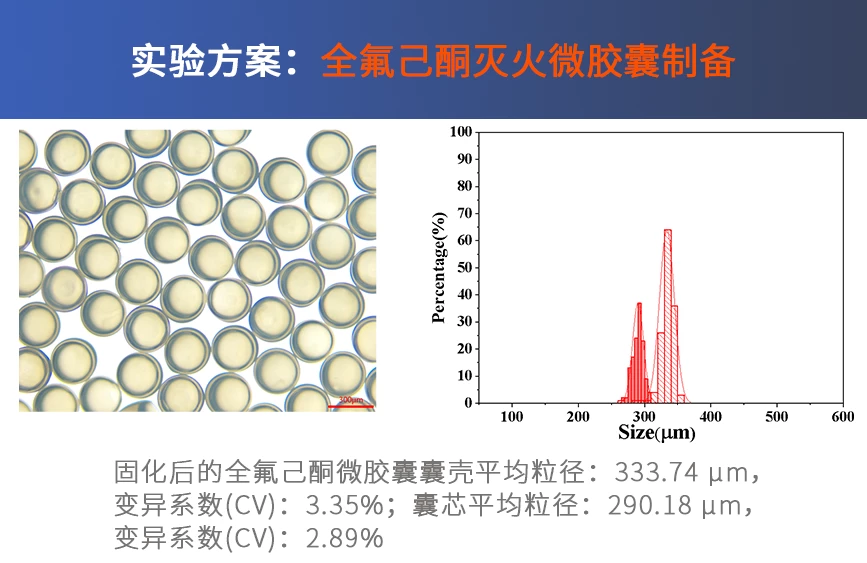


![图5. 基于微流控的冻融循环制备PVA微球[5]](https://cnfluidiclab.oss-cn-shanghai.aliyuncs.com/wp-content/uploads/2025/08/pic-5.png)



